Le ribosome cartographié en 3D
© F. Weis, P. Bron, E. Giudice, J.-P. Rolland, D. Thomas, B. Felden, R. Gillet, 2010.
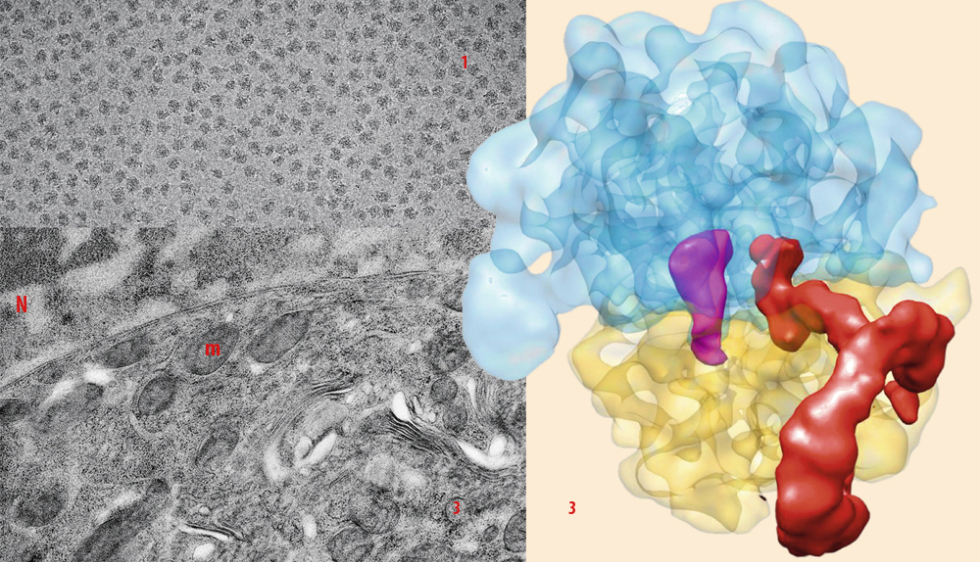
2 - Carte tridimensionnelle d’un ribosome bactérien bloqué, générée à partir des données de cryomicroscopie de 138000 particules différentes.
La densité attribuée au complexe de sauvetage (ARNtm-SmpB) est en rouge, en violet l’ARN de transfert bloqué, en jaune la petite sous-unité ribosomique et en bleu la grande.
© F. Weis, P. Bron, E. Giudice, J.-P. Rolland, D. Thomas, B. Felden, R. Gillet, 2010.
3 - Détail d’une coupe de cellule humaine cryofixée par congélation haute pression (HPF) et observée en microscopie électronique à transmission. (m) mitochondries (N) Noyau
© N. Cougot, A. Cavalier, D. Thomas, R. Gillet, 2010.
Principale cible des antibiotiques, le ribosome livre les secrets de sa morphologie à des spécialistes en imagerie.
Le prix Nobel de chimie a été décerné l’année dernière à trois chercheurs qui en ont fait la cartographie grâce à la cristallographie aux rayons X(1) : le ribosome, cette usine de fabrication des protéines longtemps schématisée sous la forme d’une “patatoïde”, livre désormais une image un peu plus précise et sophistiquée. À Rennes, le laboratoire Structure et dynamique des macromolécules(2) est à l’origine du développement d’une autre technique d’observation : la cryomicroscopie électronique.
Le regain d’intérêt des chercheurs pour cette machinerie moléculaire vient du fait qu’elle est la cible de la moitié des antibiotiques utilisés pour lutter contre les bactéries. C’est un moyen très efficace, la bactérie meurt rapidement, mais des phénomènes de résistance à certains traitements se développent. Par ailleurs, il faut pouvoir cibler les ribosomes bactériens sans endommager ceux de la cellule hôte, c’est-à-dire les nôtres !
La dynamique du ribosome
L’imagerie permet d’avoir accès à des différences morphologiques. Moins précise que la cristallographie aux rayons X qui permet de descendre à l’échelle atomique, mais qui est difficile à mettre en œuvre sur des grosses molécules (un million d’atomes pour le ribosome !), la cryomicroscopie électronique permet d’observer un nombre important de ribosomes dans des états différents et donc de comprendre leur dynamique. Visualiser, par exemple, des résistances, comprendre comment la bactérie se sort d’un mauvais pas. Quand le ribosome est bloqué, une molécule sentinelle (un ARN transfert messager) repère le blocage, se fixe sur le ribosome puis le débloque. « Nous pouvons visualiser toutes ces étapes. L’idée est ensuite de trouver de nouvelles cibles pour contourner la résistance », explique Reynald Gillet, le directeur du laboratoire. Une publication est en cours sur ce sujet(3).
L’équipe rennaise travaille aussi à améliorer la technique. En refroidissant rapidement et sous haute pression la cellule entière – et non plus des ribosomes purifiés –, il est possible de les observer dans un état très proche de leur environnement naturel(4). « J’ai l’impression du reporter animalier qui a photographié et décrit pendant des années et dans les moindres détails des ours polaires (les ribosomes) au parc zoologique et qui, aujourd’hui, commence le reportage sur la banquise ! Nul doute que l’environnement glaciaire et les autres animaux entourant notre ours polaire nous permettront de le regarder sous un angle complètement nouveau, de visualiser sa naissance (ses précurseurs) ainsi que sa vie en famille (les polyribosomes). »
Encore confidentielle
La technologie est encore confinée au laboratoire, mais Rennes essaime son savoir-faire : un des étudiants encadrés par Reynald Gillet vient de soutenir sa thèse sur le sujet. Les ribosomes n’ont qu’à bien se tenir !
Un ARN suspect en observation à Rennes
Les chercheurs du laboratoire de biochimie pharmaceutique de Brice Felden lui ont trouvé un rôle de suspect dans le déclenchement de la virulence du staphylocoque doré (lire article ci-dessus). Il a donc été transféré dans le laboratoire de biologie structurale de Reynald Gillet pour être mis en observation avec les techniques de cryomicroscopie électronique (lire article ci-contre), où l’on cherche à percer ses liens avec le ribosome du staphylocoque doré. Les premières images prêtes à confondre cet ARN sont en cours d’analyse. L’affaire, menée dans le cadre d’un projet financé par l’ANR(5), par les deux laboratoires rennais et une équipe parisienne, dure depuis un an, dans un contexte très compétitif !
(1) Venkatraman Ramakrishnan, Tom Steitz et Ada Yonath. Relire dans Sciences Ouest n°270 - novembre 2009 sur www.espace-sciences.org/ magazine.
(2) UMR 6026 CNRS/Université de Rennes 1
(3) Weis et al., tmRNA-SmpB : a journey to the centre of the ribosome. EMBO J. 2010 Oct 15.
(4) Relire “Ce que je cherche” avec Annie Cavalier, membre du laboratoire, dans Sciences Ouest n°260 - décembre 2008 sur www.espace-sciences.org/magazine.
(5) ANR : Agence nationale de la recherche.
Reynald Gille
Tél. 02 23 23 45 07
reynald.gillet [at] univ-rennes1.fr (reynald[dot]gillet[at]univ-rennes1[dot]fr)
TOUT LE DOSSIER
du magazine Sciences Ouest






